Ácido ribonucleico
O ácido ribonucléico ou RNA (em inglês, RNA para ácido ribonucléico ) é um ácido nucléico presente em praticamente todos os seres vivos e também em alguns vírus . O RNA é quimicamente muito próximo ao DNA e também é geralmente sintetizado em células a partir de um modelo de DNA do qual é uma cópia. As células usam o RNA em particular como um transportador intermediário para genes para sintetizar as proteínas de que precisam. O RNA pode cumprir muitas outras funções e, em particular, intervir nas reações químicas do metabolismo celular .
Quimicamente, o RNA é um polímero linear composto por uma cadeia de nucleotídeos . Cada nucleotídeo contém um grupo fosfato , um açúcar ( ribose ) e uma base nitrogenada (ou base nucleica). Os nucleotídeos estão ligados uns aos outros por ligações fosfodiéster . Existem quatro bases nucleicas encontradas no RNA: adenina , guanina , citosina e uracila .
O RNA tem muitas semelhanças com o DNA , mas com algumas diferenças importantes: do ponto de vista estrutural, o RNA contém resíduos de ribose onde o DNA contém desoxirribose , o que torna o RNA quimicamente menos estável; além disso, a timina no DNA é substituída por uracila, que tem as mesmas propriedades básicas de emparelhamento da adenina. Funcionalmente, o RNA é mais frequentemente encontrado em células em uma forma de fita simples , isto é, fita simples, enquanto o DNA está presente como duas fitas complementares formando uma dupla hélice . Finalmente, as moléculas de RNA presentes nas células são mais curtas do que o DNA do genoma , seu tamanho variando de algumas dezenas a alguns milhares de nucleotídeos, contra alguns milhões a alguns bilhões de nucleotídeos para o ácido desoxirribonucléico (DNA).
Na célula, o RNA é produzido pela transcrição do DNA (localizado no núcleo dos eucariotos). O RNA é, portanto, uma cópia de uma região de uma das fitas do DNA. As enzimas que fazem a cópia DNA → RNA são chamadas de RNA polimerases . Os RNAs assim produzidos podem ter três tipos principais de funções: podem ser portadores da informação genética de um ou mais genes que codificam proteínas (fala-se então de RNA mensageiro ), podem adotar uma estrutura secundária e terciária estável e desempenhar funções catalíticas ( por exemplo, RNA ribossômico ), eles podem finalmente servir como um guia ou matriz para funções catalíticas desempenhadas por fatores de proteína (que é o caso de microRNAs, por exemplo ).
Estrutura de RNA
Ribonucleotídeos
Estrutura químicaO RNA é um ácido nucléico , ou seja, uma molécula formada por uma cadeia ( polímero ) de nucleotídeos . Cada unidade de nucleotídeo do RNA consiste em uma pentose , ribose , cujos átomos de carbono são numerados de 1 ′ a 5 ′, uma base de nitrogênio variável, ou base nucleica , e um grupo fosfato . A nucleobase é conectada por um átomo de nitrogênio ao carbono um da ribose; e o grupo fosfato do nucleotídeo está ligado ao carbono 5 '. Os nucleotídeos ligam-se uns aos outros por meio de grupos fosfato, com o grupo fosfato de um nucleotídeo (ligado ao carbono 5 ') através de ligações fosfodiéster nos carbonos 3' do próximo nucleotídeo.
As quatro bases principais do RNA, e as únicas usadas no RNA de transferência, são adenina (nota A), uracila (nota U), citosina (nota C) e guanina (nota G). Em comparação com o DNA, a timina no DNA é substituída por uracila no RNA. A diferença entre essas duas bases é a substituição de um grupo metil na posição 5 da timina por um átomo de hidrogênio no uracila. Esta modificação estrutural não altera as propriedades de emparelhamento com a adenina.
A ribozima , particularmente o RNA ribossômico e o RNA de transferência, incluem outros nucleotídeos modificados, mais de cem foram identificados.
EstereoquímicaEstruturalmente, a presença de um átomo de oxigénio na posição 2 'da ribose influências a conformação do anel de furanose de ribose. Este heterociclo de cinco átomos não é planar, o que leva a dois principais conformadores de açúcar, chamados C2′-endo e C3′-endo. No RNA, que possui um átomo de oxigênio na posição 2 ', a posição C3′-endo é favorecida, o que modifica profundamente a estrutura das hélices duplas que compreendem as fitas de RNA. Esses duplexes de RNA formam uma hélice do tipo A, diferente daquela observada predominantemente no DNA convencional, que é uma hélice do tipo B, onde a desoxirribose está na conformação C2-endo.
Dupla hélice de RNA
A hélice do tipo A que o RNA adota quando forma um duplex tem propriedades geométricas bastante diferentes das da hélice do tipo B. Em primeiro lugar, o número de pares de bases por volta da hélice é 11 em vez de 10 para o DNA da forma B. O plano dos pares de bases não é mais perpendicular ao eixo da hélice, mas forma um ângulo de cerca de 75 ° com ele. Isso resulta em um deslocamento do eixo da hélice que não passa mais pelo centro do emparelhamento das bases, mas dentro do sulco maior. Isso induz um aumento no diâmetro da hélice que vai de cerca de 20 Å para o DNA na forma B a cerca de 26 Å para o RNA na forma A. Finalmente, a geometria das duas ranhuras é profundamente afetada: a ranhura principal torna-se muito acessível, enquanto a pequena ranhura se torna muito profunda, estreita e comprimida. Isso tem um impacto sobre como o RNA de fita dupla pode interagir com as proteínas, uma vez que a estreiteza do sulco menor é uma barreira para a acessibilidade dos ligantes proteicos.
Estrutura in vitro
A maioria dos RNAs de ocorrência natural estão presentes na forma de fita simples (fita simples) na célula, ao contrário do DNA, que está na forma de fita dupla emparelhada. As fitas de RNA dobram-se principalmente sobre si mesmas, formando uma estrutura intramolecular que pode ser muito estável e muito compacta. A base dessa estrutura é a formação de pares internos, entre bases complementares ( A com U , G com C e, às vezes, G com U ). A descrição dos pares internos entre as bases de um RNA é chamada de estrutura secundária . Esta estrutura secundária pode ser completada por interações remotas que então definem uma estrutura tridimensional ou estrutura terciária .
A formação da estrutura do RNA é muitas vezes dependente das condições físico-químicas circundantes e, em particular, da presença, na solução , de cátions divalentes , como o íon magnésio Mg 2+ . Esses cátions interagem com os grupos fosfato da espinha dorsal e estabilizam a estrutura, em particular protegendo a repulsão eletrostática entre as cargas negativas desses fosfatos.
A estrutura terciária do RNA é a base da riqueza de suas funções e, em particular, de sua capacidade de catalisar as reações químicas ( ribozimas ).
Estrutura secundária
A estrutura secundária de um RNA é a descrição de todos os pares internos dentro de uma molécula de fita simples. Este conjunto de emparelhamentos induz uma topologia particular, composta de regiões helicoidais (hastes) e regiões desemparelhadas (loops). Por extensão, a estrutura secundária também cobre a descrição desta topologia.
A formação de estruturas secundárias dentro de um RNA de fita simples resulta da existência de regiões contendo sequências palindrômicas , que podem emparelhar para formar localmente uma estrutura de dupla hélice. Por exemplo, se o RNA contém as seguintes duas sequências : --GUGCCACG ------ CGUGGCAC-- , estas formam uma sequência palindrômica, os nucleotídeos do segundo segmento sendo complementares aos do primeiro, após inversão de seu sentido de ler; esses dois segmentos podem então emparelhar de forma antiparalela para formar uma região localmente duplex. A região entre os dois segmentos forma um "laço" conectando os dois fios emparelhados, formando esse emparelhamento uma "haste". Isso é então referido como uma estrutura em "grampo de cabelo" ou em forma de haste .
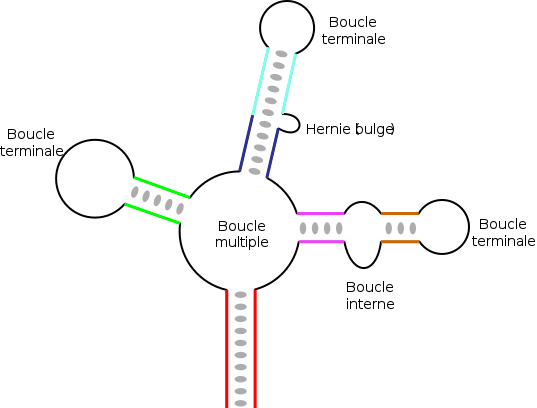
Em RNAs de maior comprimento, podem existir estruturas mais complexas que resultam do emparelhamento de várias regiões complementares ou palindrômicas . Dependendo da forma como essas diferentes regiões são "aninhadas", obtém-se vários elementos topológicos , com hastes ou regiões emparelhadas e vários tipos de loops:
- os laços terminais , localizados na extremidade de uma haste;
- os laços internos , que conectam as duas hastes;
- os múltiplos laços que conectam três ou mais hastes e formam a estrutura dos pontos de conexão;
- a hérnia ( protuberância inglesa ) ou alças laterais que estão em um dos dois fios de uma hélice. A continuidade da hélice geralmente não é afetada e as bases permanecem empilhadas coaxialmente, em cada lado da hérnia.
Nem sempre existe uma única estrutura estável para uma determinada sequência e acontece que certos RNAs podem adotar várias conformações alternativas dependendo da ligação de um ligante ( proteína , molécula pequena, etc.) ou de condições físico-químicas ( força iônica , pH ). Em geral, a formação ou fusão da estrutura secundária de um RNA pode ser seguida por medições espectroscópicas . Assim, por exemplo, a absorção no ultravioleta das bases do RNA é maior no estado não dobrado do que no estado dobrado (fenômeno da hipercromia ).
Estrutura terciária
Emparelhamentos não canônicosAlém da topologia de loops e hélices compostas de pares de bases padrão, um RNA pode adotar uma estrutura tridimensional compacta, ou estrutura terciária , como uma proteína . Dentro desta estrutura, as hélices canônicas são completadas por emparelhamentos não canônicos, ou seja, distintos dos emparelhamentos clássicos, de Watson - Crick ( A = U e G ≡ C ) e oscilação ( oscilação , G = U ). Uma ampla variedade de tais pares foi observada em estruturas tridimensionais de RNA resolvidas por cristalografia de raios-X ou ressonância magnética nuclear . Há, por exemplo, pares de bases de Hoogsteen e “cortados” emparelhamentos . Existem também interações base - ribose , especialmente com 2 ' hidroxila , que pode formar ligações de hidrogênio . Uma nomenclatura sistemática de todas essas interações foi proposta por Eric Westhof e seus colaboradores. Mais de 150 tipos de pares foram observados e agrupados em doze grandes famílias. Esses pares não canônicos sempre envolvem ligações de hidrogênio entre as bases, que são coplanares , como nos pares Watson-Crick.
Interações de longa distânciaEmparelhamentos canônicos ou não canônicos podem ocorrer entre regiões distantes da estrutura secundária, muitas vezes localizadas em loops, o que possibilita a estabilização de um dobramento compacto da estrutura.
Algumas dessas interações não canônicas de longa distância incluem:
- os pseudonós , estruturas formadas pela interação de uma alça com uma região localizada fora da haste que a delimita;
- o triplex de fita , que ocorre quando uma região de fita simples é inserida no sulco principal de uma região helicoidal;
- as interações transceptor tetraloop : interações hiperestáveis entre loops de quatro nucleotídeos (tétraboucles) e estruturas duplex ou quase-duplex.
Diferenças entre DNA e RNA
As principais diferenças entre as duas moléculas são:
- RNA para açúcar a ribose onde o DNA desoxirribose ;
- a base uracila cumpre no RNA a função desempenhada pela timina no DNA;
- O RNA geralmente existe na forma de fita simples (fita simples), exceto em alguns vírus , como reovírus, onde existe como RNA de fita dupla , enquanto o DNA é de fita dupla (fita dupla) com uma estrutura de dupla hélice;
- O RNA é curto: de algumas dezenas a alguns milhares de nucleotídeos para o RNA celular (mRNA ou RNA estruturado), contra alguns milhões a alguns bilhões (três bilhões em humanos ) no DNA que constitui o genoma da célula.
As três primeiras diferenças dão ao RNA muito menos estabilidade do que o DNA:
- O grupo ribose possui uma hidroxila na posição 2 ', que está ausente na desoxirribose do DNA . Esta função 2′-OH tem múltiplas implicações para a estrutura do RNA. Em primeiro lugar, no nível químico, essa função do álcool torna o RNA sensível à hidrólise alcalina. A presença dos dois oxigênio cis nas posições 2 ′ e 3 ′ torna possível a ciclização do fosfato nas posições 2 ′ e 3 very, que ocorre muito rapidamente quando uma base rasga o próton do 2′-OH. Esta ciclização do nucleotídeo causa um corte da cadeia ribose-fosfato e libera 5'-OH e 2 ', 3' terminais de fosfato cíclicos ;

- O uracil é menos caro em energia para produzir para os organismos vivos do que a timina, uma vez que requer uma etapa de síntese a menos do que a metilação pela timidilato sintase . A presença de timina no DNA permite que a célula detecte danos espontâneos à citosina, que é sensível à oxidação . A desaminação espontânea da citosina na presença de oxigênio converte-a em uracila. A presença de desoxiuridina no DNA é anormal, uma vez que o desoxirribonucleotídeo complementar da desoxiadenosina é a timidina . Com essa distinção entre timina e uracila por meio de um grupo metil, o sistema básico de reparo excisional pode detectar e corrigir o defeito. No RNA, a desaminação das citosinas produz uracilos e não é reparada. Como o RNA tem uma vida útil muito mais curta do que o DNA (da ordem de um minuto), ele é degradado e reciclado;
- se uma única fita de RNA é danificada, a lesão não é reparada e o dano é irreversível; por outro lado, se uma das duas fitas de DNA for danificada, a célula pode usar a informação carregada pela fita complementar intacta para reparar a lesão .
Do ponto de vista evolutivo, certos elementos nos permitem pensar que o RNA seria anterior ao DNA como portador de informação genética, o que explicaria suas funções mais extensas e sua generalização. O DNA teria surgido mais tarde e suplantado o RNA apenas para o papel de armazenamento de longo prazo, devido à sua maior estabilidade.
Síntese de RNA a partir de DNA
A síntese de uma molécula de RNA a partir do DNA é chamada de transcrição . É um processo complexo que envolve uma enzima da família da RNA polimerase , bem como proteínas associadas. Os diferentes estágios desta síntese são iniciação, alongamento e término. O processo de síntese de RNA é marcadamente diferente em organismos procarióticos e em células eucarióticas . Finalmente, após a transcrição real, o RNA pode sofrer uma série de modificações pós-transcricionais como parte de um processo de maturação durante o qual sua sequência e estrutura química podem ser modificadas (veja abaixo).
Iniciação
O início da transcrição de um RNA por uma RNA polimerase ocorre no nível de uma sequência específica do DNA, chamada de promotor . Este promotor compreende um ou mais elementos de sequência conservada aos quais proteínas específicas, os fatores de transcrição , são geralmente fixados . Logo a montante do local de iniciação da transcrição, o elemento proximal é geralmente rico em nucleotídeos T e A e, portanto, é chamado de caixa TATÁ em eucariotos ou caixa de Pribnow em bactérias. Fatores de transcrição promovem o recrutamento de RNA polimerase para o promotor e a abertura do duplex de DNA. O que é chamado de bolha de transcrição é então formado com o DNA aberto, uma das fitas (o molde) é hibridizada com o RNA que está sendo sintetizado.
Alongamento
Uma vez que a RNA polimerase é fixada no promotor e a bolha de transcrição formada, ela sintetiza os primeiros nucleotídeos de maneira estática, sem deixar a sequência do promotor. Os fatores de transcrição são interrompidos e a RNA polimerase torna-se processual. Em seguida, ele transcreve o RNA na direção 5 'para 3' , usando uma das duas fitas de DNA como molde e trifosfatos de ribonucleotídeo ( ATP , GTP , CTP e UTP ) como precursores.
In vivo , em Escherichia coli , a taxa de extensão da RNA polimerase é de cerca de 50 a 90 nucleotídeos por segundo.
Terminação
A terminação da transcrição do RNA ocorre de acordo com mecanismos completamente diferentes em bactérias e em eucariotos .
Nas bactérias, o principal mecanismo de terminação envolve uma estrutura particular de RNA, o terminador , que consiste em uma alça de haste estável seguida por uma série de resíduos de uridina (U). Quando a RNA polimerase sintetiza essa sequência, o dobramento do bastão de RNA faz com que a polimerase pare. O RNA, que não está mais emparelhado com o DNA molde, exceto por uma série de pares de UA fracos, se separa, sem a intervenção de outros fatores proteicos. A terminação também pode ser feita por meio da intervenção de um fator proteico específico, o fator Rho.
Em eucariotos, a terminação da transcrição pela RNA polimerase II é acoplada à poliadenilação . Dois complexos de proteínas, CPSF (en) e CStF (en) reconhecem poliadenilação (5′-AAUAAA-3 ′) e sinais de clivagem de RNA. Eles clivam o RNA, induzem o desprendimento da DNA polimerase e recrutam a poli-A polimerase que adiciona a cauda poli (A) (veja abaixo).
Maturação
O processamento de RNA compreende um conjunto de modificações pós-transcricionais observadas principalmente em eucariotos e desempenham um papel importante no destino do RNA maturado. As principais modificações são a adição de um boné 5 ′, a poliadenilação 3 ′, o splicing , a introdução de modificações químicas no nível da base ou da ribose e, finalmente, a edição .
BonéO cap , ou 5 '-cap em inglês, é um nucleotídeo modificado que é adicionado à extremidade 5' do RNA mensageiro em células eucarióticas . Consiste em um resíduo de guanosina metilada ligada por uma ligação trifosfato 5'-5 'ao primeiro nucleotídeo transcrito pela RNA polimerase . Essa modificação é introduzida no núcleo da célula, pela ação sucessiva de várias enzimas: polinucleotídeo 5'-fosfatase , RNA guanililtransferase , metiltransferases .
O cap desempenha várias funções: aumenta a estabilidade do RNA protegendo-o da degradação pelas exonucleases 5′-3 3 e também permite o recrutamento de fatores de iniciação da tradução necessários para a ligação do ribossomo aos RNAs. Mensageiros celulares. A tampa é, portanto, essencial para a tradução da maioria dos mRNAs.
PoliadenilaçãoA poliadenilação é a adição de uma extensão à extremidade 3 'do RNA composta exclusivamente por ribonucleotídeos do tipo adenosina (A). Por esse motivo, a extensão é chamada de polocauda (A) . Embora composta de nucleotídeos padrão, esta cauda poli (A) é adicionada pós-transcricionalmente por uma enzima específica chamada poli (A) polimerase e não é codificada no DNA genômico . A cauda poli (A) é encontrada principalmente no final dos RNAs mensageiros . Nos eucariotos, a poliadenilação dos mRNAs é necessária para sua tradução pelo ribossomo e participa de sua estabilização. A cauda poli (A) é em particular reconhecida por PABP ( proteína de ligação poli (A) , " proteína de ligação poli (A) ").
Em bactérias e em certas mitocôndrias , a poliadenilação do RNA é, ao contrário, um sinal de degradação.
SplicingO splicing é uma modificação pós-transcricional que envolve a remoção de íntrons e a sutura dos exons no mRNA e em algum RNA estruturado, como o tRNA. Encontrados em organismos eucarióticos, os íntrons são segmentos de RNA que são codificados no genoma e transcritos em RNA precursor, mas que são removidos do produto final. Na maioria dos casos, esse processo envolve uma maquinaria complexa específica chamada spliceossomo . O splicing ocorre no núcleo das células eucarióticas, antes da exportação do RNA maturado para o citoplasma.
Nucleotídeos modificadosApós sua transcrição pela RNA polimerase, alguns RNAs sofrem modificações químicas sob a ação de enzimas específicas. Os principais RNAs que estão sofrendo mudanças são os RNAs de transferência e os RNAs ribossômicos . Também pode ser considerado que as metilação envolvidas na síntese do cap são modificações de nucleotídeos particulares. No caso geral, as modificações podem ser relativas à base ou à ribose . As principais modificações encontradas são:
- na ribose: O- metilação da posição 2'-OH;
- na base :
- isomerização de uridinas que dá pseudouridinas ,
- metilação, seja em átomos de carbono ( ribotimidina ) ou em átomos de nitrogênio ( 7-metilguanosina ...),
- uma redução , que converte uridinas em di- hidrouridinas ,
- tiolações,
- modificações mais complexas ( prenilação , treonil-carbamoilação, etc.).
Em RNAs de transferência, a introdução de nucleotídeos modificados contribui para aumentar a estabilidade das moléculas.
EditandoA edição do RNA consiste em uma modificação da sequência do ácido ribonucleico, subsequente à transcrição pela RNA polimerase. No final do processo de edição, a sequência do RNA é, portanto, diferente da do DNA. As alterações feitas podem ser a modificação de uma base, a substituição de uma base ou a adição de uma ou mais bases. Essas modificações são realizadas por enzimas que atuam no RNA, como a citidina desaminase , que convertem quimicamente resíduos de citidina em uridina .
Função na célula
Nas células, os RNAs cumprem quatro funções distintas e complementares:
- suporte temporário à informação genética : o RNA mensageiro cumpre esse papel, é utilizado pela célula para transmitir a informação correspondente a um determinado gene fora do núcleo, para então sintetizar proteínas a partir dessa informação;
- catalisador enzimático : como as proteínas , os RNAs podem se dobrar em três dimensões para formar estruturas complexas. Essas estruturas permitem que certos RNAs se comportem como enzimas , isso é chamado de ribozima . O ribossomo , a ribonuclease P e alguns íntrons são ribozimas . Existem argumentos indiretos que indicam que a maquinaria de splicing do RNA mensageiro (o spliceossomo ) também é uma ribozima, embora isso ainda não tenha sido formalmente demonstrado;
- guia para enzimas : alguns RNAs são usados como co-fatores por proteínas para permitir seu direcionamento para sequências específicas. Entre eles estão pequenos RNAs nucleolares (snoRNAs), que orientam as enzimas modificadoras do RNA ribossômico , o RNA telomérico, que é um cofator da telomerase , a enzima que forma as extremidades dos cromossomos, ou os RNAs interferentes ;
- reguladores da expressão gênica : certos RNAs não codificantes desempenham um papel na repressão da expressão de certos genes ou grupos de genes. Este é o caso, por exemplo, de RNAs antisense que emparelham com um RNA alvo e bloqueiam a tradução do mesmo pelo ribossomo.
Uma classe especial de RNA, os RNAs de transferência, são encontrados na interface de muitas dessas funções, guiando os aminoácidos durante a tradução .
Finalmente, o genoma de alguns vírus é composto exclusivamente de RNA e não de DNA. Este é particularmente o caso dos vírus da gripe , AIDS , hepatite C , poliomielite e Ebola . Dependendo do caso, a replicação desses vírus pode passar por um intermediário de DNA ( retrovírus ), mas também pode ser feita diretamente de RNA para RNA.
O RNA é uma molécula versátil, o que levou Walter Gilbert , co-inventor do sequenciamento do DNA , a propor em 1986 a suposição de que o RNA seria a mais antiga de todas as macromoléculas biológicas. Essa teoria, conhecida como hipótese do mundo do RNA (“ hipótese do mundo do RNA ”), permite superar um paradoxo do ovo e da galinha que surge quando se busca saber quais das proteínas ( catalisadores ) e quais do DNA (genéticas informações) apareceu primeiro. Nesse modelo, o RNA, capaz de combinar os dois tipos de funções ao mesmo tempo, seria o precursor universal.
RNA mensageiro
A informação genética contida no DNA não é usada diretamente pela célula para sintetizar a proteína . Para isso, ele usa cópias transitórias de informações genéticas que são RNAs mensageiros ou mRNAs. Cada RNA mensageiro carrega um ou, às vezes, vários cistrons , ou seja, as instruções para formar uma única proteína. Portanto, corresponde à cópia de apenas um dos genes do genoma (fala-se então de mRNA monocistrônico) ou às vezes de alguns ( mRNA policistrônico ).
O RNA mensageiro contém uma cópia de apenas uma das duas fitas de DNA, a codificadora, e não a sequência complementar. Em comparação com a sequência do gene contido no DNA do genoma, a do mRNA correspondente pode conter modificações, em particular devido ao splicing (ver acima) que elimina as regiões não codificantes . O RNA mensageiro sintetizado no núcleo da célula é exportado para o citoplasma para ser traduzido em proteína. Ao contrário do DNA, que é uma molécula perene, presente ao longo da vida da célula, os RNAs mensageiros têm uma vida útil limitada, de alguns minutos a algumas horas, após a qual são degradados e reciclados.
Um RNA mensageiro tem três regiões distintas: uma região 5 'não traduzida chamada 5'-UTR, localizada a montante do cistron ou cistrons que ele carrega; uma região de codificação correspondente a este ou esses cistrons; e, finalmente, uma região 3 'não traduzida chamada 3' UTR. Os RNAs mensageiros são traduzidos em proteínas pelos ribossomos . A região 5 'não traduzida geralmente contém os sinais translacionais que permitem o recrutamento do ribossomo no cistron. O processo de tradução também envolve a transferência de RNAs que fornecem ao ribossomo os aminoácidos necessários para a biossíntese de proteínas . Dentro do ribossomo, por meio de seu anticódon , os tRNAs emparelham sucessivamente com os tripletos de bases, ou códons , da sequência de mRNA. Quando o emparelhamento códon-anticódon está correto, o ribossomo adiciona o aminoácido transportado pelo tRNA à proteína que está sendo sintetizada. As correspondências entre códons e aminoácidos constituem o código genético .
A função dos RNAs mensageiros é múltipla. Por um lado, permitem preservar o molde original do DNA, que não é usado diretamente para a tradução, a célula trabalhando apenas na cópia que é o mRNA. Acima de tudo, a existência de RNAs mensageiros fornece à célula um mecanismo crucial para regular o ciclo de produção de proteínas do genoma. A necessidade celular de uma determinada proteína pode variar de acordo com o ambiente, o tipo de célula, o estágio de desenvolvimento. A síntese de proteínas deve, portanto, ser ativada ou interrompida dependendo das condições celulares. A regulação da transcrição do DNA em mRNA atende a essa necessidade e é controlada por fatores de transcrição específicos que atuam nos promotores dos genes-alvo. Quando a quantidade de uma determinada proteína é suficiente, a transcrição do mRNA é inibida, é gradualmente degradada e a produção da proteína cessa. Portanto, é importante que o mRNA seja uma molécula transitória, para poder realizar essa regulação essencial.
RNA de transferência
O RNA de transferência , ou tRNA, é um RNA curto, com cerca de 70 a 100 ribonucleotídeos envolvidos no direcionamento dos aminoácidos para o ribossomo durante a tradução .
Os RNAs de transferência têm uma estrutura de folha de trevo característica, que consiste em quatro hastes emparelhadas. Uma dessas hastes é terminada por uma alça que contém o anticódon , o tripleto de nucleotídeos que emparelha com o códon durante a tradução de um mRNA pelo ribossomo . Na outra extremidade, o tRNA carrega o aminoácido correspondente ligado por uma ligação éster em sua extremidade 3'-OH. Essa esterificação é catalisada por enzimas específicas, as aminoacil-tRNA sintetases . Em três dimensões, a estrutura da folha do trevo se dobra em forma de "L", com o anticódon em uma extremidade e o aminoácido esterificado na outra.
Todas as células vivas contêm um conjunto de diferentes tRNAs contendo diferentes aminoácidos e capazes de ler diferentes códons.
Os RNAs de transferência são às vezes chamados de "adaptadores" entre a sequência genética e a sequência da proteína . Foi Francis Crick quem propôs a existência desses adaptadores, antes mesmo de sua descoberta em 1958.
RNAs catalíticos ou ribozimas
A descoberta do RNA com capacidade catalisadora foi feita na década de 1980, principalmente pela equipe de Thomas Cech , que trabalhou nos íntrons do gene do RNA ribossomal do protozoário ciliado Tetrahymena , e do Sidney Altman , que estudou a ribonuclease P , a enzima de maturação do tRNA . Cech e Altman receberam o Prêmio Nobel de Química em 1989 por esta descoberta.
Em ambos os casos, o RNA sozinho é capaz de catalisar uma clivagem específica (clivagem) ou reação de transesterificação na ausência de proteína . Esses RNAs catalíticos são chamados de ribozimas porque são enzimas compostas de ácido ribonucléico. No caso do íntron Tetrahymena , ele é um autossplicing , sendo o íntron seu próprio substrato , enquanto a ribonuclease P é uma enzima que atua em trans , em múltiplos substratos.
Desde essas descobertas iniciais, outras ribozimas naturais foram identificadas:
- RNAs de viróides ou vírus satélite (virusóides) que são capazes de se clivar;
- hoje existem argumentos muito fortes, baseados na resolução de sua estrutura 3D, para afirmar que o próprio ribossomo , a ribonucleoproteína complexa responsável pela tradução do mRNA em proteínas , é uma ribozima. Os dois sítios ativos do ribossomo, o centro de decodificação na subunidade pequena e o centro da peptidiltransferase que forma as ligações peptídicas , são de fato exclusivamente compostos por segmentos de RNA ribossômico;
- o spliceossomo , que catalisa o splicing de mRNAs citoplasmáticos de eucariotos , provavelmente também é uma ribozima;
- certos riboswitches , que são regiões reguladoras estruturadas transportadas por RNAs mensageiros, têm atividade de clivagem catalítica na presença de um ligante ;
- Por fim, existem as ribozimas sintéticas, isoladas por métodos evolutivos in vitro , como a técnica SELEX . RNAs catalíticos sintéticos capazes de catalisar uma ampla variedade de reações químicas e ligar uma ampla variedade de ligantes foram isolados, o que tem sido interpretado como um argumento a favor da hipótese do mundo do RNA . Esses RNAs sintéticos às vezes são chamados de aptâmeros , uma vez que são "adequados" para realizar uma determinada tarefa.
Em geral, em todas essas ribozimas, é o seu dobramento específico que lhes permite realizar o reconhecimento do seu substrato e a catálise, como no caso das enzimas protéicas.
Guias de RNA
Os RNAs guias são RNAs que se combinam com enzimas protéicas e servem para guiar sua ação em RNAs ou DNAs de sequências complementares . O RNA guia emparelha-se com o ácido nucleico substrato e ajuda a direcionar a atividade da enzima. Vários tipos foram identificados:
- o pequeno RNA nucleolar , ou snoARN : no nucléolo das células eucarióticas, eles dirigem a ação das enzimas modificadoras do RNA ribossômico, particularmente 2'-O-metilação por snoARN C / D e por pseudouridilações H / ACA snoRNAs. Esse mecanismo permite que a célula modifique especificamente várias posições do rRNA, com uma única enzima usando diferentes snoRNAs como guias. SnoRNAs são frequentemente codificados por sequências intrônicas ;
- os microRNAs também são RNAs guias envolvidos no processo de interferência de RNA : associados a um complexo proteico denominado RISC ( complexo de silenciamento induzido por RNA ), esses pequenos RNAs causam uma deterioração do mRNA alvo ao qual se acasalam, seja uma repressão de sua tradução ;
- TERC ( telomerase RNA component ), a subunidade de RNA da telomerase : este RNA estruturado está associado à transcriptase reversa que sintetiza telômeros , as extremidades dos cromossomos . Ele contém uma sequência que serve como substrato para a telomerase sintetizar DNA telomérico de sequência complementar. Portanto, guia a atividade da enzima, mas servindo como um modelo, ao invés de formar um emparelhamento com o substrato;
- os lincARN presentes em mamíferos são grandes RNAs não codificadores intergênicos, mas transcritos como mRNA pela RNA polimerase II. Seu comprimento permite que eles adotem uma estrutura tridimensional complexa. Essas estruturas permitem sua interação com diferentes cofatores transcricionais, como hnRNP-K ou PRC2 (principalmente inibidores da transcrição). Esses complexos são então guiados pelo lincRNA nas sequências regulatórias dos genes para inibir sua expressão. A ligação dos lincRNAs com o DNA envolveria um emparelhamento das bases do RNA com as bases do DNA correspondentes após o desemparelhamento da dupla hélice do DNA, ou mesmo a formação da tripla hélice DNA-DNA-RNA.
Reguladores de RNA
Certos RNAs desempenham um papel de reguladores diretos da expressão gênica. Este é, em particular, o caso de RNAs não codificantes possuindo regiões complementares aos RNAs mensageiros celulares e que podem, portanto, emparelhar com eles para formar localmente uma fita dupla de RNA. Esses RNAs antisense podem ser derivados do mesmo locus genético que seu RNA alvo, por transcrição da fita complementar, que é então referida como RNA cis- regulador. Eles também podem ser derivadas a partir da transcrição de uma outra região do genoma, que são então trans reguladoras RNAs .
O emparelhamento do RNA regulador com seu RNA mensageiro alvo pode atuar na capacidade deste último de ser traduzido pelo ribossomo ou em sua estabilidade, o que leva a uma regulação da tradução do (s) gene (s) transportado (s) pelo RNA. . Em bactérias, existem muitos exemplos de RNA anti-sentido cis - ou trans- regulador que bloqueia o local de início da tradução. Por exemplo, o gene que codifica a porina OmpF é regulado por um RNA anti-sentido denominado MicF.
Em eucariotos , também existem grandes RNAs regulatórios, que estão envolvidos em processos regulatórios epigenéticos . O exemplo mais conhecido é o do RNA Xist em mamíferos. Isso inativa não um gene, mas um cromossomo inteiro. O Xist cobre um dos dois cromossomos X de cada célula em indivíduos do sexo feminino, que se torna inativo. Assim, apenas um dos dois cromossomos do par XX está ativo, o que permite ter o mesmo nível de expressão dos genes transportados pelo cromossomo X que em indivíduos do sexo masculino, que possuem apenas um. A inativação de X é um processo aleatório, que pode levar à expressão de diferentes fenótipos por diferentes células, na mesma fêmea. É o caso, por exemplo, da cor da pelagem dos gatos.
Usos terapêuticos e biotecnológicos
O RNA é usado hoje em uma série de aplicações em biologia molecular, em particular graças ao processo de interferência de RNA , que consiste na introdução em células eucarióticas de pequenos fragmentos de RNA de fita dupla chamados " pequenos RNAs interferentes ". Com cerca de vinte pares de bases de comprimento, esses pequenos RNAs interferentes (pRNAs) são usados por uma maquinaria celular capaz de degradar mRNAs de uma maneira específica. Apenas os mRNAs contendo uma sequência correspondente à do pRNAi são degradados, o que permite diminuir seletivamente a expressão de uma determinada proteína. Esta abordagem tecnológica é muito mais simples e rápida do que o estabelecimento de linhas de mouse inativadas ( knock-out ) e é chamada de knock-down .
Estão previstas tentativas de usar esta técnica para fins terapêuticos, por exemplo, visando genes virais para combater infecções, ou oncogenes , no caso de cânceres. No entanto, eles requerem a estabilização dos pequenos RNAs interferentes (pRNAi) para evitar sua degradação pelas ribonucleases e direcionar sua ação para as células em questão.
Histórico
Os ácidos nucléicos foram descobertos em 1868 por Friedrich Miescher . Miescher chamou a nova substância de "nucleína" porque foi encontrada no núcleo das células. A presença de ácidos nucléicos no citoplasma das leveduras foi identificada em 1939 e sua natureza ribonucléica foi estabelecida, ao contrário dos cromossomos que continham DNA com desoxirriboses.
Por volta de 1940 , o biólogo belga Jean Brachet estudava moléculas até então pouco caracterizadas, ainda denominadas na época “ácidos timomonucleico e zimonucléico” (DNA e RNA respectivamente). Ele descobre que o ácido timonucleico é um componente dos cromossomos e que é sintetizado quando as células se dividem após a fertilização . Ele destaca a existência de ácidos zimonucléicos (RNA) em todos os tipos de células: no núcleo , nucléolo e citoplasma de todas as células (embora se pensasse na época que essas moléculas eram características de células vegetais e eucariotos inferiores, como leveduras ). Finalmente, mostra que esses ácidos são particularmente abundantes nas células (mais particularmente no ergastoplasma ), que são muito ativas em termos de síntese protéica . Os fundamentos fundamentais da biologia molecular foram estabelecidos. O ano era 1940. No período do pós-guerra, Brachet foi acompanhado pelo biólogo molecular belga Raymond Jeener, que participaria ativamente da pesquisa sobre o papel do RNA na biossíntese de proteínas .
No final da década de 1950, Severo Ochoa conseguiu sintetizar moléculas de RNA in vitro por meio de uma enzima específica, o polinucleotídeo fosforilase, que possibilitou o estudo das propriedades físicas e químicas do RNA.
O papel do RNA como um “mensageiro” intermediário entre a informação genética contida no DNA e as proteínas foi proposto em 1960 por Jacques Monod e François Jacob após uma discussão com Sydney Brenner e Francis Crick . A demonstração da existência do RNA mensageiro foi feita por François Gros . A partir daí, a decifração do código genético foi feita por Marshall Nirenberg na primeira metade da década de 1960. Para isso, utilizou RNAs sintéticos de sequência de nucleotídeos conhecida, cujas propriedades codificadoras estudou.
Os ribossomos foram observados pela primeira vez pelo biólogo belga Albert Claude no início da década de 1940. Usando técnicas de fracionamento subcelular e microscopia eletrônica , ele revelou "pequenas partículas" de natureza ribonucleoproteica, presentes em todos os tipos de células, células vivas. Ele os chamou de "microssomas", mais tarde renomeados ribossomos.
A estrutura secundária dos tRNAs foi estabelecida por Robert Holley , que conseguiu purificar e analisar a sequência de tRNA específica da alanina em 1964. Esse foi um grande avanço na compreensão da decifração da mensagem genética transmitida por eles, os RNAs mensageiros. A estrutura tridimensional de um tRNA foi resolvida em 1974, de forma independente, pelas equipes de Aaron Klug e Alexander Rich, mostrando pela primeira vez a estrutura complexa de um RNA. A existência de propriedades catalíticas de RNAs foi estabelecida de forma independente por Sidney Altman e Tom Cech em 1982, na ribonuclease P por um lado e nos íntrons autossplicantes por outro. A resolução da estrutura das subunidades individuais do ribossomo em 2000 pelas equipes de Tom Steitz , Ada Yonath e Venki Ramakrishnan , depois a de todo o ribossomo pela equipe de Harry Noller em 2001, constituiu um avanço essencial na compreensão da o mecanismo central da biologia que é a tradução de mRNAs em proteínas. Além disso, permitiu mostrar, entre outras coisas, que o ribossomo também era uma ribozima.
Durante a década de 1970, Timothy Leary , em sua obra The Politics of Ecstasy , viu no RNA a promessa de uma futura modificação da consciência (possivelmente por meio de novas drogas e / ou exercícios espirituais), incluindo seria um componente que aumentaria as capacidades de aprendizagem de alguém que se envolveria em tais experiências.
Hipótese do mundo de RNA
A hipótese do mundo do RNA é uma hipótese segundo a qual o RNA é o precursor de todas as macromoléculas biológicas e, particularmente, do DNA e das proteínas que teriam permitido em um ambiente abiótico (caracterizado por uma química pré - biótica que é parcialmente hipotética) o aparecimento dos primeiros seres vivos células, isto é, formando um compartimento, e compreendendo subsistemas de informação e metabólicos.
No contexto do estudo das origens da vida , esta hipótese permite uma explicação do aparecimento de diferentes funções biológicas através da constituição de certos blocos biomoleculares a partir de intermediários prebióticos plausíveis e moléculas baseadas no carbono. Foi demonstrado em 2009 pela equipe de John Sutherland que precursores plausíveis de ribonucleotídeos, aminoácidos e lipídios podem ser obtidos por homologação redutiva do cianeto de hidrogênio e alguns de seus derivados. Cada um dos subsistemas celulares conhecidos poderia, portanto, ser explicado pela química do carbono, com reações catalisadas pela luz ultravioleta a priori muito presente antes do aparecimento da camada de ozônio , a partir do sulfeto de hidrogênio como agente redutor. O próprio ciclo fotoredutivo pode ser acelerado pelo cobre [Cu (I) -Cu (II)].
Notas e referências
- (em) Rebecca K. Montange e Robert T. Batey , " Structure of the S-adenosylmethionine riboswitch mRNA Regulation element " , Nature , vol. 441, n o 7097,29 de junho de 2006, p. 1172-1175 ( PMID 16810258 , leia online )
- H. Lodish , A. Berk , P. Matsudaira , CA Kaiser , M. Krieger , MP Scott , L. Zipursky e J. Darnell , Biologia molecular da célula , Bruxelas, de Boeck,2005, 3 e ed. ( ISBN 978-2804148027 )
- (en) Wolfram Saenger , Principles of Nucleic Acid Structure , Springer,1984( ISBN 0-387-90762-9 )
- (em) janeiro Barciszewski e Brian Frederick Carl Clark , RNA bioquímica e biotecnologia , Springer,1999, 73–87 p. ( ISBN 0-7923-5862-7 , OCLC 52403776 )
- Modificação e edição do Rna. Henri Grosjean, Rob Benne. ASM Press, 1998.
- Marie-Christine Maurel, Anne-Lise Haenni: O mundo do RNA: hipóteses, fatos e resultados experimentais.
- (in) Mr. Sudaralingam , " Estereoquímica dos ácidos nucléicos e seus constituintes. 4. Conformações permitidas e preferidas de nucleosídeos, mono-, di-, tri-, tetrafosfatos, ácidos nucleicos e polinucleotídeos de nucleosídeos ” , Biopolymers , vol. 7, N o 6,1969, p. 821-860 ( ler online )
- (em) R. Langridge e PJ Gomatos , " The Structure of RNA. O RNA do reovírus e o RNA de transferência têm estruturas tridimensionais semelhantes, que diferem do DNA. » , Science , vol. 141, n o 4,1963, p. 694-698 ( PMID 13928677 )
- (en) HR Drew , RM Wing , T. Tanako , C Broka , S Tanaka , K Itakura e RE Dickerson , " Structure of a B-DNA dodecamer: conformation and dynamics. » , Proc. Natl. Acad. Sci. EUA , vol. 78, n o 4,Abril de 1981, p. 2179-2183 ( PMID 6941276 , leia online )
- (in) Peter S. Klosterman , Sapan A. Shah e Thomas A. Steitz , " Crystal structure of two plasmid copy control RNA duplexes related: An 18 pares de bases duplexes em 1,20 A de resolução e 19 pares de bases duplexes em 1,55 A de resolução . ” , Biochemistry , vol. 38, n o 45,1999, p. 14784-14792 ( PMID 10555960 , DOI 10.1021 / bi9912793 , ler online )
- (en) JM Rosenberg , NC Seeman , JJ Kim , FL Suddath , HB Nicholas e A. Rich , " Double Helix at atomic resolution. » , Nature , vol. 243, n o 5403,1973, p. 150-154 ( PMID 4706285 , leia online )
- (em) RO Day , NC Seeman , MJ Rosenberg e A. Rich , " A Crystalline Fragment of the Double Helix: The Structure of the dinucleoside phosphate Guanylyl-3 ', 5'-Cytidine. » , Proc. Natl. Acad. Sci. EUA , vol. 70, n o 3,Março de 1973, p. 849-853 ( PMID 4514996 , leia online )
- (em) Alexander Rich e David R. Davies , " A new two stranded helical structure: Polyadenylic polyuridylic acid and acid " , J. Am. Chem. Soc. , vol. 78, n o 14,1956, p. 3548-3549 ( DOI 10.1021 / ja01595a086 , leia online )
- (em) OF Draper , " Protein-RNA Recognition " , Annu. Rev. Biochem. , vol. 64,1995, p. 593-620 ( PMID 7574494 , leia online )
- (em) SA Woodson , " íons metálicos e dobramento de RNA: um tópico altamente carregado com um futuro dinâmico " , Curr. Opiniões Chem. Biol. , vol. 9, n o 2Abril de 2005, p. 104-9 ( PMID 15811793 , leia online )
- (em) P. Doty , H. Boedtker , JR Fresco , R. Haselkorn e M. Litt , " Secondary Structure in Ribonucleic Acids " , Proc. Natl. Acad. Sci. EUA , vol. 45, n o 4,1959, p. 482-499 ( PMID 16590404 )
- F. Dardel e F. Képès , Bioinformática: genômica e pós-genômica , Editions de l'École Polytechnique,2002, 153-180 p. ( ISBN 978-2730209274 )
- (em) AM Michelson , " Hyperchromicity and nucleic ácidos. » , Nature , vol. 182, n o 4648,1958, p. 1502-1503 ( PMID 13613306 )
- (em) K. Hoogsteen , " The crystal and molecular structure of hidrogênio-bonded complex entre-1 e 9-methylthymine methyladenine. » , Acta Cryst. , vol. 16,1963, p. 907-916 ( DOI 10.1107 / S0365110X63002437 )
- (em) HA Heus e A. Pardi , " Structural features That give rise to the anormal estabilidade of RNA hairpins Containing NRMA loops. » , Science , vol. 253, n o 50161991, p. 191-194 ( PMID 1712983 )
- (in) NB Leontis e E. Westhof , " The non-Watson-Crick base pairs and their isostericity associated matrices. ” , Nucleic Acids Res. , vol. 30, n o 16,2002, p. 3497-3531 ( PMID 12177293 )
- (em) DW Staple e SE Butcher , " Pseudoknots: RNA structure with Diverse Functions. » , PloS Biol. , vol. 3, n o 6,2005, e213 ( PMID 15941360 , leia online )
- (em) Mr. Costa e F. Michel , " Frequent use of the Sami tertiary pattern by self-Folding RNAs " , EMBO J. , Vol. 14,1995, p. 1276–1285 ( PMID 7720718 , leia online )
- (em) PJ Gomatos e I. Tamm , " The Second structure of reovirus RNA " , Proc. Natl. Acad. Sci. EUA , vol. 49, n o 5,1963, p. 707-714 ( PMID 16591092 )
- (em) R. Markham e JD Smith , " The Structure of Ribonucleic Acids 1. Cyclic nucleotides andproduced by ribonuclease by alcaline hydrolysis " , Biochem. J. , vol. 52, n o 4,1952, p. 552-557 ( PMID 13018277 , leia online )
- Walter Gilbert, " O Mundo do RNA ", Nature 319, 1986, p. 618
- Harper's Biochemistry, Harold A Harper, A Harold
- (em) ST Smale e JT Kadonaga , " The RNA polimerase core promoter " , Ann. Rev. Biochem. , vol. 72,2003, p. 449-479 ( PMID 12651739 , leia online )
- (em) RP Lifton , L. Goldberg , RW Karp e DS Hogness , " A organização dos genes das histonas em Drosophila melanogaster: implicações funcionais e evolutivas " , Cold Spring Harb. Legal. Quant. Biol. , vol. 42,1978, p. 1047-1051 ( PMID 98.262 , lido online )
- (em) D. Pribnow , " Nucleotide sequence of RNA polimerase binding year website at an early T7 promoter " , Proc. Natl. Acad. Sci. EUA , vol. 72,1975, p. 784-788 ( PMID 1093168 , leia online )
- (em) BA Hamkalo e OL Miller , " electronmicroscopy of genetic activity " , Annu. Rev. Biochem. , vol. 42,1973, p. 376-396 ( PMID 4581229 )
- (em) WR McClure e Y. Chow , " The kinetics and processivity of nucleic acid polimerases " , Methods Enzymol. , vol. 64,1980, p. 277-297 ( PMID 6990186 , leia online )
- (em) H. Bremer e PP Dennis, "Modulação da composição química e outros parâmetros da célula pela taxa de crescimento" em Neidhardt FC, R Curtiss, III, JL Ingraham ECC Lin KB Low, B Magasanik WS Reznikoff, M Riley, M Schaechter e HE Umbarger, Escherichia coli e Salmonella typhimurium Cellular and Molecular Biology , Washington, DC, ASM Press,1996( ISBN 0-914826-89-1 , leia online ) , p. 1553-1569
- (em) S. Adhya and Mr. Gottesman , " Control of transcription termination " , Annu. Rev. Biochem. , vol. 47,1978, p. 967-996 ( PMID 354508 , leia online )
- (em) MS Ciampi , " terminadores de transcrição dependente de Rho e determinação " , Microbiologia , vol. 152,2006, p. 2515-2528 ( PMID 16946247 , ler online )
- (in) Mr. Edmonds , " Uma história de sequências poli A: da formação aos fatores à função " , Prog. Nucleic Acid res. Mol. Biol. , vol. 71,2002, p. 285-389 ( PMID 12102557 , leia online )
- (en) AK Banerjee , " cap 5'-terminal structure in Eucaryotic messenger ribonucleic acid " , Microbiol. Rev , vol. 44, n o 21980, p. 175-205 ( PMID 6247631 , leia online )
- (em) Mr. Edmonds e R. Abrams , " polinucleotide biosynthesis: formação de uma sequência de unidades adenilato de adenosina trifosfato por uma enzima de núcleos do timo " , J. Biol. Chem. , vol. 235,1960, p. 1142-1149 ( PMID 13819354 , leia online )
- (em) Mr. Dreyfus e P. Regnier , " The poly (A) tail of mRNAs: bodyguard in ekaryotes, bactéria in scavenger " , Cell , vol. 111,2002, p. 611-613 ( PMID 12464173 , leia online )
- (em) JP Staley e C. Guthrie , " Mechanical devices of the spliceosome: motors, clocks, springs, and things " , Cell , vol. 92,1998, p. 315-326 ( PMID 9476892 , leia online )
- (em) WE Cohn , " Pseudouridine, a carbon-carbon linked ribonucleoside in ribonucleic acidos: isolamento, estrutura e características químicas " , Journal of Biological Chemistry , vol. 235,1960, p. 1488-1498 ( PMID 13811056 , leia online )
- (em) JA Kowalak JJ Dalluge , JA McCloskey e KO Stetter , " O papel da modificação pós-transcricional na estabilização do RNA de transferência de hipertermofílico. ” , Biochemistry , vol. 28,1994, p. 7869-7876 ( PMID 7516708 )
- (en) S. Valadkhan , A. Mohammadi , Y. Jaladat e S. Geisler , “ pequenos RNAs nucleares livres de proteínas catalisam uma reação de splicing em duas etapas. » , Proc. Natl. Acad. Sci. EUA , vol. 106,2009, p. 11901-11906 ( PMID 19549866 )
- (em) S. Brenner , F. Jacob e M. Meselson , " Um intermediário instável transportando informações de genes para ribossomos para síntese de proteínas. » , Nature , vol. 190,1961, p. 576-581
- (em) BN Ames e RG Martin , " Biochemical aspect of genetics: The operon. » , Annu. Rev. Biochem. , vol. 33,1964, p. 235-258 ( PMID 14268834 )
- (em) C. Yanofsky , " Establishing the Nature of the triplet genetic code. ” , Cell , vol. 128,2007, p. 815-818 ( PMID 17350564 )
- (em) Huijing Shi e Peter B. Moore , " A estrutura cristalina do tRNA da fenilalanina de levedura com resolução de 1,93 Å: A classic revisited structure " , RNA , Vol. 6, n o 8, agosto de 2000, p. 1091-1105 ( PMID 10943889 , PMCID 1369984 , DOI 10.1017 / S1355838200000364 , ler online )
- (em) MB Hoagland , ML Stephenson , JF Scott , HI Hecht e PC Zamecnik , " A soluble ribonucleic acid intermediário em síntese de proteína " , J. Biol. Chem. , vol. 231,1958, p. 241-257 ( PMID 13538965 )
- (em) RW Holley , J. Apgar , GA Everett , JT Madison , M. Marquisee , SH Merrill , JR Penswick e A. Zamir , " Structure of a ribonucleic acid " , Science , vol. 147,1965, p. 1462-1465 ( PMID 14263761 )
- (en) JD Robertus , JE Ladner , JT Finch , D. Rhodes , RS Brown , BF Clark e A. Klug , “ Structure of yeast fenilalanine tRNA at 3 A resolution. » , Nature , vol. 250,1974, p. 546–551 ( PMID 4602655 ), (en) SH Kim , FL Suddath , GJ Quigley , A. McPherson , JL Sussman , AH Wang , NC Seeman e A. Rich , “ Estrutura terciária tridimensional do RNA de transferência de fenilalanina de levedura. » , Science , vol. 250,1974, p. 546–551 ( PMID 4601792 )
- (em) Francis H. Crick , " We protein synth " , Symp. Soc. Exp. Biol. , vol. 12,58, p. 138-163 ( PMID 13580867 , leia online )
- (en) K. Kruger , PJ Grabowski , AJ Zaug , J. Sands , DE Gottschling e TR Cech , “ ARN de auto-ligao: autoexcision e autocyclization da sequência de ARN ribossómico de Tetrahymena interveniente. ” , Cell , vol. 31,1982, p. 147-157 ( PMID 6297745 )
- (en) C. Guerrier-Takada , K. Gardiner , T. Marsh , N. Pace e S. Altman , “ A porção de RNA da ribonuclease P é a subunidade catalítica da enzima. ” , Cell , vol. 35,1983, p. 849-857 ( PMID 6197186 )
- (in) AC Forster e Davies , " Characterization of self-cleavage of viroid and virusoid RNAs. " , Methods Enzymol. , vol. 181,1990, p. 583-607 ( PMID 2199768 )
- (em) TR Cech , " Biologia Estrutural. O ribossomo é uma ribozima. » , Science , vol. 289,2000, p. 878-879 ( PMID 10960319 )
- (em) I Barrick , KA Corbino , WC Winkler , A. Nahvi , Sr. Mandal , J. Collins , Sr. Lee , A. Roth , N. Sudarsan I. Jona , JK Wickiser e RR Breaker , " New RNA motifs sugerem um escopo expandido para riboswitches no controle genético bacteriano. » , Proc. Natl. Acad. Sci. EUA , vol. 101,2004, p. 6421-6426 ( PMID 15096624 )
-
(in) AD Ellington e JW Szostak , " Seleção in vitro de moléculas de RNA que ligantes específicos. » , Nature , vol. 346,1990, p. 818-822 ( PMID 1697402 )
(en) C. Tuerk e L. Gold , “ Systematic evolution of ligands by exponential enriquecimento: RNA ligands to bacteriófago T4 DNA polimerase. » , Science , vol. 249,1990, p. 505-510 ( PMID 2200121 ) - (em) Z. Kiss-László , Y. Henry , JP Bachellerie , Sr. Caizergues Ferrer e T. Kiss , " Site-specific ribose methylation of RNA preribosomal: a novel function for small nucleolar RNAs. ” , Cell , vol. 85,1996, p. 1077-1088 ( PMID 8674114 )
- (en) J. Liu , " Novel intron-encoded small nucleolar RNAs. ” , Cell , vol. 75,1993, p. 403-405 ( PMID 8221882 )
- (in) B. Sollner-Webb , " Controle da síntese de proteínas e degradação de mRNA por microRNAs. " , Curr. Opin. Cell Biol. , vol. 20,2008, p. 214-221 ( PMID 18329869 )
- (em) D. Shippen-Lentz e EH Blackburn , " Functional evidence for an RNA template in telomerase. » , Science , vol. 247,1990, p. 546-552 ( PMID 1689074 )
- (em) Mr. Huarte , T. Jacks e JL Rinn , " A Large intergenic noncoding RNA Induced by p53 medeia Global Gene Repression in the p53 Response " , Cell , vol. 142,2010, p. 409-419 ( PMID 20673990 )
- (in) EG Wagner e RW Simons , " Antisense RNA control in bactéria, phages and plasmids. » , Annu. Rev. Microbiol. , vol. 48,1994, p. 713-742 ( PMID 3685996 )
- (en) E. Heard , " Recent advance in X-chromosome inactivation. " , Curr. Opin. Cell Biol. , vol. 16,2004, p. 247-255 ( PMID 15145348 )
- (em) A. Fire , S. Xu , Sr. Montgomery , S. Kostas S. Driver e C. Mello , " Potent and specific geneticinterference by double-stranded RNA in Caenorhabditis elegans . » , Nature , vol. 391, 1998, p. 806-811
- Claude Hélène , “ As promessas do RNA terapêutico = Interferência genética pelo RNA. », Le Concours Médical , vol. 124,2002, p. 2550-2552
- (em) R Dahm , " Friedrich Miescher e a descoberta do DNA ' , Dev. Biol. , vol. 278, n o 22005, p. 274–88 ( PMID 15680349 , DOI 10.1016 / j.ydbio.2004.11.028 )
- (em) T. Caspersson e J. Schultz , " Pentose nucleotides in the cytoplasm of growth tecidos " , Nature , vol. 143,1939, p. 602-3 ( DOI 10.1038 / 143602c0 )
- https://www.ulb.ac.be/ibmm/histoire.html
- (em) F. Jacob e J. Monod , " Genetic Regulation Mechanisms in the synth of proteínas. ” , Journal of Molecular Biology , vol. 3,1961, p. 318-356 ( PMID 13718526 )
- François Jacob , A estátua interior , Gallimard, col. "Folio" ( n O 2156)1990, 438 p. ( ISBN 978-2-070-38246-0 e 2-070-38246-X , observe BnF n o FRBNF35085459 )
- (em) F. Gros , H. Hiatt , W. Gilbert , CG Kurland , RW Risebrough e JD Watson , " Unstable ribonucleic acid Revealed by pulse labeling of Escherichia coli " , Nature , vol. 190, 1961, p. 581-585 ( PMID 13708983 )
- (em) A. Claude , " A constituição do protoplasma " , Ciência , vol. 97, n o 25251943, p. 451-456 ( PMID 17789864 , DOI 10.1126 / science.97.2525.451 )
- (em) N. Ban , P. Nissen J. Hansen , PB Moore , TA Steitz , " The complete atomic structure of the ribosomal subunit wide at 2.4 A resolution. » , Science , vol. 289, 2000, p. 905-920 ( PMID 10937989 )
- (em) F. Schluenzen , A. Tocilj R. Zarivach , J. Harms , Sr. Gluehmann , D. Janell , A. Bashan , H. Bartels , I. Agmon , F. Franceschi e A. Yonath , " Structure of functionally subunidade ribossômica pequena ativada com resolução de 3,3 angstroms. ” , Cell , vol. 102, 2000, p. 615-623 ( PMID 11007480 )
- (em) OF Brodersen , " Structure of the 30S ribosomal subunit. » , Nature , vol. 407,2000, p. 1143-1154 ( PMID 11014182 )
- (en) MM Yusupov , GZ Yusupova , A. Baucom , K. Lieberman , TN Earnest , JH Cate e HF Noller , " Crystal structure of the ribosome at 5.5 A resolution. » , Nature , vol. 292,2001, p. 883-896 ( PMID 11283358 )
- A política do êxtase, Timothy Leary, 1974, Ed. Fayard, Paris, p. 115-120
- Bhavesh H. Patel, Claudia Percivalle, Dougal J. Ritson, Colm D. Duffy e John D. Sutherland (2015), Origens comuns de RNA, proteínas e precursores lipídicos em um protometabolismo cianossulfídico ; Nature Chemistry ( resumo )
Veja também
Artigos relacionados
- Lista de tipos de RNA
- Ácido ribonucléico mensageiro
- Ribossomo
- Ribozima
- RNA de fita dupla
- microRNA
- RNA de interferência
- Lista de abreviações de biologia celular e molecular
- fita transcrita
- RNA-Seq
- Lista de ácidos
links externos
- RNA World no Fritz Lipman Institute Coleção de links no “mundo RNA” (estruturas, sequências, ferramentas, publicações)
